肝了好几天终于把想要做的部分都弄好了,来交个overdue的作业
简介
TaichiMD 是基于 Taichi 编程语言,以分子动力学为核心的实时交互式粒子模拟引擎。其特性和目标包括:
- 通过 Taichi 编程语言搭建计算机图形学到和分子模拟/计算化学的桥梁
- 通过GPU加速实现美观,实时,可交互的粒子模拟
- 为新的模拟算法的快速实现和可微分模拟提供平台
最终效果
512个丙烷分子的分子动力学模拟,使用我们组的TraPPE-UA力场加上简谐形式的化学键伸缩(其实我一直没加每个粒子的质量,所以不是完全准确的)

(gif被压得完全变色了,原图看这儿)
Taichi 的“99行代码冰雪奇缘”经典示例 MPM99,使用32768个MPM粒子和64 * 64 * 64网格
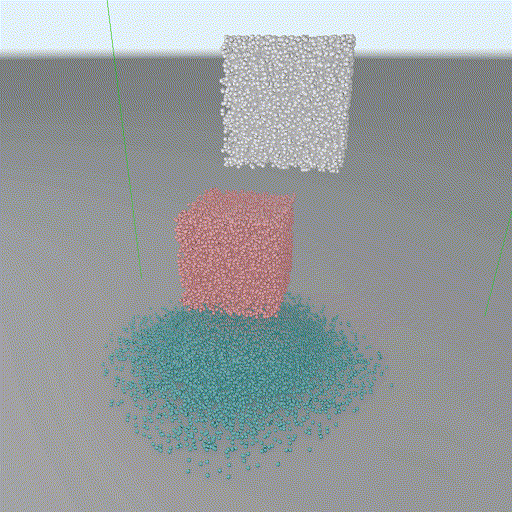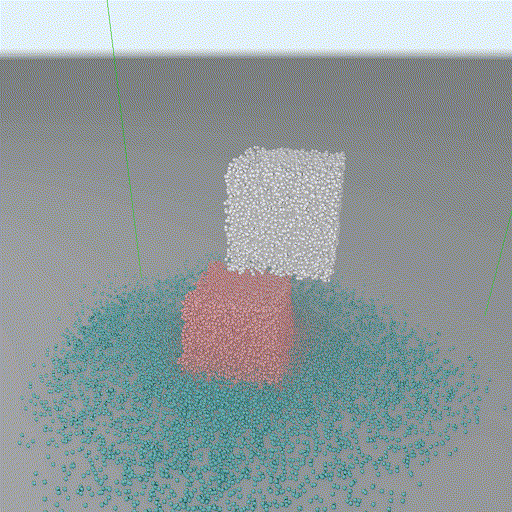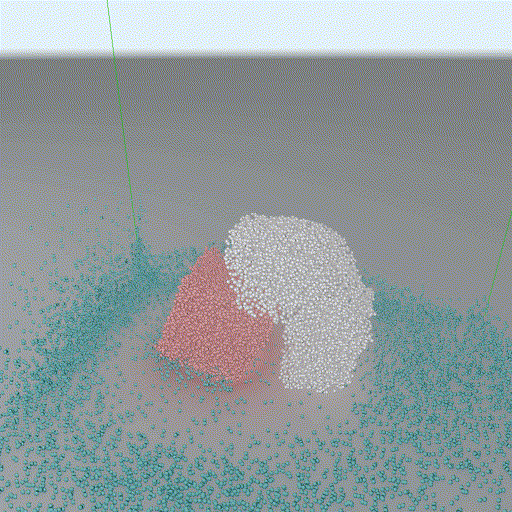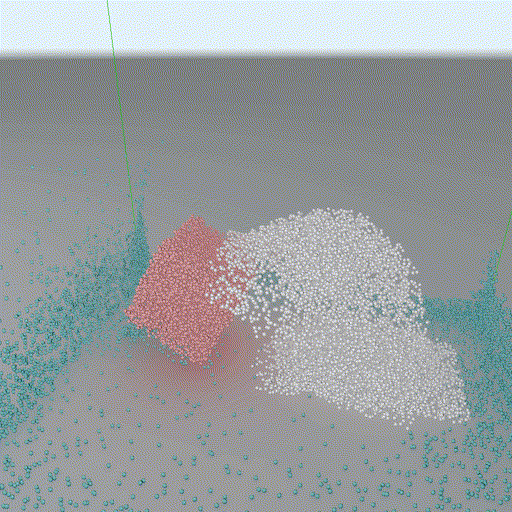
代码示例
现在版本的TaichiMD在作业1的基础上增加的大量的功能,比如多组分混合物的模拟,化学键弯曲的bending potential,以及一系列基于网格的方法,包括Neighbor List加速模拟,PIC和APIC。最后,MPM99的三种材料模拟被压缩到了48行:
import taichi as ti
from taichimd import Simulation, MDRenderer, DIM
from taichimd.grid import APIC, QuadraticKernel
from taichimd.integrator import ForwardEulerIntegrator as FE
MDRenderer.radius = 0.2
class MPMGrid(APIC):
@ti.func
def affine(self, i):
self.system.F[i] = (ti.Matrix.identity(ti.f32, DIM) + self.system.dt * self.system.C[i]) @ self.system.F[i]
h = 0.3 if self.system.type[i] == 1 else ti.exp(10 * (1.0 - self.system.Jp[i]))
la = self.lambda_0 * h
U, sig, V = ti.svd(self.system.F[i])
J = 1.0
for d in ti.static(range(DIM)):
new_sig = min(max(sig[d, d], 1 - 2.5e-2), 1 + 4.5e-3) if self.system.type[i] == 2 else sig[d, d]
self.system.Jp[i] *= sig[d, d] / new_sig
sig[d, d] = new_sig
J *= new_sig
if self.system.type[i] == 0: # Reset deformation gradient to avoid numerical instability
self.system.F[i] = ti.Matrix.identity(ti.f32, DIM)
self.system.F[i][0, 0] = J
elif self.system.type[i] == 2:
self.system.F[i] = U @ sig @ V.T() # Reconstruct elastic deformation gradient after plasticity
stress = ti.Matrix.identity(ti.f32, DIM) * la * J * (J - 1)
if self.system.type[i] != 0:
stress += 2 * self.mu_0 * h * (self.system.F[i] - U @ V.T()) @ self.system.F[i].T()
stress = (-self.system.dt * self.vol * 4 * self.inv_dx * self.inv_dx) * stress
return stress + self.mass * self.system.C[i]
ti.init(arch=ti.cuda, device_memory_GB=3)
dt = 2e-4
n_particles, n_grid = 32768, 64
p_vol, p_rho = (0.5 / n_grid)**2, 1
mpmgrid = MPMGrid(QuadraticKernel(), n_grid, mass=p_vol * p_rho, gravity=50)
mpmgrid.vol = p_vol
E, nu = 0.1e4, 0.2 # Young's modulus and Poisson's ratio
mpmgrid.mu_0, mpmgrid.lambda_0 = E / (2 * (1 + nu)), E * nu / ((1+nu) * (1 - 2 * nu)) # Lame parameters
sim = Simulation(n_particles, integrator=FE(dt, False), grid=mpmgrid)
sim.add_attr("F", dims=(DIM, DIM), dtype=ti.f32) # affine velocity field, deformation gradient
sim.add_attr("Jp", dims=(), dtype=ti.f32) # plastic deformation
sim.gui.set_colors([[6/255, 133/255, 135/255], [237/255, 85/255, 89/255], [238/255, 238/255, 240/255]]) # mpm99 colors
sim.init_random(center=(0.4, 0.15, 0.7), length=0.2, start=0, end=n_particles//3, inittype=0)
sim.init_random(center=(0.4, 0.45, 0.7), length=0.2, start=n_particles//3, end=2*n_particles//3, inittype=1)
sim.init_random(center=(0.5, 0.75, 0.7), length=0.2, start=2*n_particles//3, end=n_particles, inittype=2)
sim.build()
sim.velocity.fill(0); sim.F.fill(((1,0,0),(0,1,0),(0,0,1))); sim.C.fill(0); sim.Jp.fill(1)
sim.run(irender=10, pause=True)
- 前五行都是import,还有设定粒子渲染的半径比例。之后我们定义了一个MPMGrid的类,MLS-MPM和APIC关系很近,所以我们就直接继承自带的APIC网格类。MLS-MPM和APIC的区别是每个粒子的affine matrix如何计算,而P2G和G2P的其他部分可以完全一样,所以就只重写affine()这个方法,抄一遍mpm99例子的代码即可。
- 定义完MPMGrid之后就进行初始化和设定mpm99对应的各种参数。然后我们新建一个Simulation对象作为我们的模拟体系,这个体系使用不含力的显示积分和我们刚才写的MPM网格。
- 模拟体系看到我们使用了APIC网格的子类,会给我们自动新建每个粒子的位置、速度和affine matrix场(C[i])以及每个粒子的类型。然而为了实现mpm99我们还需要给每个粒子加入F和Jp这两个属性(
sim.add_attr(...)) - 然后我们设定每种粒子的颜色和初始化的位置,再调用
sim.build()这个方法,会触发taichi的materialize过程,并place上模拟体系中的各种粒子和网格变量。 - 在开始模拟之前我们把各种物理量设为初始值,然后用
sim.run()开始模拟。pause=True意味着模拟不会自动运行,按下空格键才会开始运行(并且编译运行时需要的各种kernel)
图形渲染
既然要做基于Taichi的交互式模拟引擎,那我们肯定需要一个完全用Taichi实现的实时渲染器。既然要实时,那么taichi例子给的各种path tracing肯定是不行的,这就得自己造轮子。好在@archibate于斌的Taichi-Three开了个好头,就可以站在巨人的肩膀上少造一些轮子。
TaichiMD是一个粒子模拟引擎,只要能渲染球形粒子(和圆柱体的化学键)就可以了。那么除了传统的三角形栅格化方法,我们就可以做一些更“直接”的解决方案,否则每个粒子是一个几百面的球,几万个粒子渲染的开销是巨大的。所以TaichiMD使用的是完全implicit的几何进行实时渲染,换句话说上面那两个GIF里面,其实一个三角形都没有。这样最直接的好处就是随粒子数量的scalability非常强,3万多个粒子的栅格化渲染在我的2080S上可以做到40–50fps,然而坏处就是因为taichi只能并行遍历每个粒子的for loop,假如1个粒子占了大半个屏幕就反而会卡了。
Taichi-Three自带只有最简单的phong lighting model,不够好看,所以我又自己写了一个Physically-based的Cook-Torrance BRDF。然而到这个时候我们还是只有direct lighting,依然不够好看(本科帮实验室里画图的,强迫症)。想要模拟indirect lighting最简单的办法是搞一个SSAO,但是粒子模拟的场景只有球体,肯定有更优雅的解法。于是我就照着GPU Gems 2里的一章最简单近似下推了个球形体系全局光(GI)的解,用Taichi实现了居然真能管用!下面请欣赏乞丐版RTX ON对比图(模拟界面内按tab打开全局光):
[全局光照关]
[全局光照开]
[全局光照关]
[全局光照开]
可以看到效果是很明显的,第一张图的红色大果冻的diffuse反光会在地面上显示,粒子的小球与小球之间的occlusion也能显示。虽然不一定比SSAO快(需要对每个fragment迭代一遍每个粒子),但这个方法有一点好,因为它是closed-form的解,所以直接渲染到frame buffer里,不需要后处理就几乎没有任何噪声。至于详细的推导过程有时间了可以写一篇文章发到我的博客上。
总结
GAMES201终于迎来了大结局。在此期间我学到了很多东西,TaichiMD也从homework 0的200行小程序到现在集粒子、网格、微观模拟、宏观模拟、实时渲染为一体的逐步成熟的项目,说这门课它是我本科和研究生加起来最有用的几个都不为过(本科学的化学所以90%课都已经没用了hhhh)。
最后这么长的帖子能你一直读到这也不容易,祝学业、工作、科研顺利!



